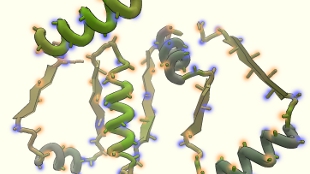
Estrutura de proteína descoberta por jogadores na Internet
O Grupo ‘The contender’ composto por pessoas de pelo menos três Continentes descobriu qual a conformação de uma proteína que intrigava a comunidade científica há dez anos. Normalmente a descoberta científica é feita entre paredes, por grupos de cientistas que trabalham em laboratórios. Desta vez a descoberta coube a um conjunto de pessoas que jogavam um jogo na internet, o Foldit. Será que a autoria das descobertas científicas do futuro vai ser assim partilhada por cidadãos?
A descoberta da conformação da proteína em causa foi publicada na prestigiada revista Nature Structural and Molecular Biology, dia 18 de Setembro de 2011. Este mistério já tinha dez anos de idade. E foi resolvido de uma forma inusitada, com um método inovador. A partir de um software, um jogo na internet, um grupo de pessoas ao brincar com a proteína que nada no seu ecrã ganharam pontos, muitos pontos. O Grupo de nome lutador ou o competidor (‘The contender’) é um dos muitos participantes no jogo online Foldit. Este jogo foi criado por David Baker com o propósito especial de ter uma ajudinha na descoberta da estrutura tridimensional de proteínas.
As proteínas têm como blocos de construção os aminoácidos, dos quais conseguimos descobrir facilmente a identidade e sequência… Mas como é que estes se arranjam na estrutura final chamada de terciária? Há sempre grupos químicos que são mais “amigos” da água enquanto outros são, pelo contrário, hidrofóbicos. Estes grupos com propriedades e tamanhos diferentes arranjam-se na sequência proteica, escondendo ao máximo os grupos hidrofóbicos, obtendo assim o estado óptimo de energia na sua estrutura final. Cada proteína tem assim no seu código um puzzle que se pode provar fácil de decifrar ou difícil de resolver.
A proteína desta descoberta é uma protease retroviral do vírus dos macacos de Mason-Pfizer e a sua estrutura era um mistério já há 10 anos. A sua descoberta pode ter um impacto no desenvolvimento de fármacos anti-retrovirais, como anti-HIV. E tudo a partir de um jogo de computador.
O Rosetta@home é um software que funciona como protector de ecrã em computadores. Enquanto alguém deixa o computador por alguns instantes, este começa a executar complexos cálculos matemáticos para desvendar estruturas de proteínas. No ecrã dançam as moléculas. Baker começou a receber e-mails de utilizadores que se queixavam da falta de interacção. Enquanto passeava com o seu amigo David Salesin pelo Monte Rainier, vulcão imponente a sudeste de Seattle, Estados Unidos, os dois reflectiam sobre este problema. Quem tinha o Rosetta@home no computador queria poder interagir com a proteína no ecrã, deslocar um braço de amino-ácidos ou empacotar a molécula numa forma mais coesa. A certa altura, Baker comentou: “Se pudéssemos só deixá-los agarrar a proteína e faze-lo eles próprios eles ficariam verdadeiramente contentes”.
Agora e enquanto brincavam com a proteína no Foldit desenhado por Baker, os “lutador” são co-autores de uma importante descoberta. Rhiju Das, que fez pós-doutoramento no laboratório de Baker, leva a questão mais além. “Será que conseguimos transformar 10,000 ou 100,000 pessoas em cidadãos cientistas que desenvolvem hipóteses, fazem experiências e depois refinam as suas hipóteses?”. Eu gostava de experimentar. E você? Quer fazer ciência?
João Cão
© 2011 - Ciência na Imprensa Regional / Ciência Viva
João Cão
Veja outros artigos deste/a autor/a.
Escreva ao autor deste texto
Ficheiros para download Jornais que já efectuaram download deste artigo

